РНК-содержащие вирусы
РНК-содержащие вирусы — вирусы, геном которых представлен рибонуклеиновой кислотой[1]. Обычно она одноцепочечная (оцРНК), но может быть и двуцепочечной (дцРНК)[2]. Наиболее серьёзными заболеваниями человека, вызываемыми РНК-вирусами, являются геморрагическая лихорадка Эбола, ТОРС, COVID-19, бешенство, простуда, грипп, гепатит C, гепатит E, лихорадка Западного Нила, полиомиелит и корь.
Международный комитет по таксономии вирусов (ICTV) относит РНК-вирусы к группам III, IV или V по системе классификации вирусов по Балтимору и не считает РНК-вирусы вирусами, имеющие промежуточную стадию жизненного цикла в виде ДНК[3]. Такие вирусы, с РНК в качестве генетического материала и имеющие промежуточную стадию жизненного цикла в виде ДНК в цикле репликации, названы ретровирусами, и выделены в группу VI классификация вирусов по Балтимору. Важные ретровирусы человека включают ВИЧ-1 и ВИЧ-2, вызывающих ВИЧ-инфекцию.
Другим термином, явно исключающий группу ретровирусов из РНК-вирусов, является термин рибовирусы[4].
Характеристики
[править | править код]Одноцепочечные РНК-вирусы и смысловая РНК
[править | править код]РНК-вирусы могут быть дополнительно классифицированы в соответствии с полярностью их РНК на одноцепочечные РНК-вирусы с отрицательной цепью[англ.] и одноцепочечные РНК-вирусы с положительной цепью[англ.], или РНК обеих типов[англ.] двухцепочечные РНК-вирусы[англ.]. Одноцепочечная (+)РНК, подобная мРНК и способная непосредственно транслироваться зараженной клеткой, называется смысловой. Антисмысловая (-)РНК вируса комплементарна мРНК и таким образом может быть превращена в (+)РНК действием РНК-зависимой РНК-полимеразой перед трансляцией. Очищенная РНК (+)РНК-содержащих вирусов сама по себе может напрямую вызывать инфекцию, хотя её инфекционность ниже, чем у полноценных вирусных частиц. Очищенная РНК (-)РНК-содержащих вирусов неинфекционна сама по себе, так как она должна транскрибироваться в (+)РНК; каждый вирион после транскрипции может быть источником нескольких смысловых цепей (+)РНК. Двухцепочечные (±)РНК вирусы напоминают (-)РНК-содержащие вирусы тем, что они также транслируют гены с отрицательной (некодирующей) цепи РНК[5].
Двуцепочечные РНК-вирусы
[править | править код]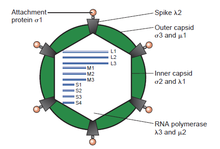
Двуцепочечные РНК-вирусы — разнообразная группа вирусов, отличающихся хозяевами (люди, животные, растения, грибы или бактерии), числом фрагментов генома (от 1 до 12 сегментов), и организацией вириона (формой капсида, количеством слоев капсида, выросты, шипы, и т. п.). Члены этой группы включают ротавирусы, всемирно известные как наиболее распространённая причина детских гастроэнтеритов, и пикорнавирусы, известный во всём мире как наиболее часто встречающийся вирус в фекалиях людей и животных с признаками диареи или без них. Вирус болезни синего языка[англ.] является экономически важным возбудителем болезни коз и овец. В прошлые годы, достигнут большой прогресс в определении атомной и субнанометровой структуры ряда ключевых вирусных белков и капсидов вирионов нескольких дцРНК-вирусов, подчёркивая существенные параллели в структуре и механизмах процессов репликации многих из этих вирусов[2].
Частота мутаций
[править | править код]РНК-вирусы имеют очень высокую частоту мутаций по-сравнению с ДНК-вирусами[6], потому что вирусная РНК-зависимая РНК-полимераза не имеет способности (3'-5'-экзонуклеазной гидролитической активности) к коррекции ошибок, как ДНК-полимеразы[7]. Это одна из причин того, почему так трудно сделать эффективную вакцину для предотвращения болезней, вызываемых РНК-вирусами.[8] Ретровирусы также имеют высокую частоту мутаций, даже несмотря на то, что их промежуточная форма генома в виде ДНК интегрируется в геном хозяина (и, следовательно, после интеграции в геном, во время репликации, подвергается корректирующей проверке ДНК-полимеразой хозяина). Высокая частота мутаций обусловлена высокой частотой ошибок во время обратной транскрипции, до стадии интеграции в геном хозяина. Ошибки при этом встраиваются в обе цепи ДНК.[9] Некоторые гены РНК-вирусов важны для их репликации и мутации в них не допустимы. Например, участок генома вируса гепатита C, кодирующий коровый белок, высоко консервативен,[10] потому что он содержит структурную РНК, вовлеченную в образование участка внутренней посадки рибосомы.[11]
Репликация
[править | править код]РНК-вирусы животных классифицированы по ICTV. Выделяют три различные группы РНК-вирусов в зависимости от структуры их генома и метода репликации:
- Двуцепочечные РНК-вирусы (группа III) содержат от одной до нескольких разных молекул РНК, каждая из которых кодирует один или несколько вирусных белков.
- Одноцепочечные (+)РНК-вирусы (группа IV) используют свой геном напрямую как мРНК, при трансляции на рибосомах хозяина в один белок, который модифицируется белками хозяина и самого вируса для образования различных белков необходимых для процесса репликации. Один из них это РНК-зависимая РНК-полимераза (РНК-репликаза), которая копирует РНК вируса переводя её в двуцепочечную форму, способную к репликации. В свою очередь дцРНК служит матрицей для синтеза новых вирусных РНК-геномов.
- Одноцепочечные (-)РНК-вирусы (группа V) должны обеспечить копирование их генома РНК-репликазой для формирования смысловой (+)нити РНК. Это означает, что вирус уже должен содержать готовый фермент РНК-репликазу вместе со своим геномом. Смысловая (+) нить РНК затем выступает в качестве мРНК, которая транслируется в белки рибосомами хозяина.
Ретровирусы (группа VI) имеют одноцепочечный РНК-геном но, в общем, не относятся к РНК-вирусам потому что они используют промежуточную стадию в виде ДНК для репликации. Обратная транскриптаза, вирусный белок, поставляемый самим вирусом, после распаковки вирусной частицы конвертирует вирусную РНК в комплементарную цепь ДНК, которая после синтеза второй цепи ДНК превращается в двуцепочечную молекулу вирусной ДНК. После этого ДНК интегрируется в геном хозяина используя вирусный белок интегразу, экспрессия закодированных генов может приводить к формированию новых вирионов.
Классификация
[править | править код]Отчасти это связано с высокой частотой мутаций, которым подвергаются эти геномы. Классификация основана главным образом на типе генома (двухцепочечный, отрицательный или положительный одноцепочечный), а также количестве и организации генов. В настоящее время известно 5 порядков и 47 семейств РНК-вирусов. Есть также много неклассифицированных видов и родов.
РНК-вирусам родственны вироиды и РНК-вирусы-сателлиты. Они не относятся к РНК-вирусам и описаны отдельно.
Изучение нескольких тысяч РНК-вирусов показало существование по крайней мере пяти главных таксонов: Levivirus и родственные группы; супергруппа Picornavirus; супергруппа Alphavirus вместе с супергруппой Flavivirus; дцРНК-вирусы; и оц(-)РНК-вирусы[12]. Группа лентивирусов является предковой для всех оставшихся групп РНК-вирусов. Следующий большой водораздел пролегает между группой пикорнавирусов и остальными вирусами. Вирусы с дцРНК-геномом похоже эволюционировали от оц(+)РНК-вирусного предка, а оц(-)РНК-вирусы возникли внутри дцРНК-вирусов. Наиболее близкородственны (-)РНК-вирусам реовирусы.
Одноцепочечные РНК-вирусы с положительной цепью
[править | править код]Это самая большая группа РНК-вирусов[13] с 30 семействами. Предпринималась попытка сгруппировать их в более крупные порядки на основе анализа их РНК-зависимой РНК-полимеразы. На сегодняшний момент это предложение не получило широкого распространения из-за сомнений в пригодности использования одного гена для классификации клады.
В предложенной классификации выделены три группы[14]:
- Пикорнаподобная группа (Picornavirata): Bymoviruses, Comoviruses, Nepoviruses, Nodaviruses, Picornaviruses, Potyviruses, Sobemoviruses и подмножество Luteoviruses (beet western yellows virus и potato leafroll virus).
- Группа, похожая на флавивирусы (Flavivirata): Carmoviruses, Dianthoviruses, Flaviviruses, Pestiviruses, Statoviruses, Tombusviruses, одноцепочечные РНК-бактериофаги, вирус гепатита C и подмножество лютеовирусов (вирус желтой карликовости ячменя).
- Группа, похожая на альфавирусы (Rubivirata): Alphaviruses, Carlaviruses, Furoviruses, Hordeiviruses, Potexviruses, Rubiviruses, Tobraviruses, Tricornaviruses, Tymoviruses, apple chlorotic leaf spot virus, beet yellows virus и вирус гепатита E.
Было предложено разделение альфа-подобной (Sindbis-подобной) супергруппы на основе нового домена, расположенного вблизи N-концов белков, участвующих в репликации вируса[15]. Предлагаемые две группы:
- Группа 'Альтовирус': Alphaviruses, Furoviruses, вирус гепатита E, Hordeiviruses, Tobamoviruses, Tobraviruses, Tricornaviruses и, возможно, Rubiviruses;
- Группа 'Типовирус': apple chlorotic leaf spot virus, Carlaviruses, Potexviruses и Tymoviruses
В будущем альфа-подобную (Sindbis-подобную) супергруппу можно будет разделить на три группы: Rubi-подобные, Tobamo-подобные, и Tymo-подобные вирусы[16].
Дополнительная работа позволила выявить пять групп РНК-вирусов с положительной цепью, содержащих 14 порядков с 31 семейством и 48 родов (из них 17 семейств с 30 родами относятся к вирусам растений)[17]. Этот анализ предполагает, что альфавирусы и флавивирусы могут быть разделены на два семейства — Togaviridae и Flaviridae, соответственно, — но выделение других таксономических единиц, таких как Pestiviruses, вирус гепатита C, Rubiviruses, вирус гепатита E и Arteriviruses, могут быть неверным. Коронавирусы и торовирусы, по-видимому, представляют собой отдельные семейства в разных порядках, а не отдельные роды того же семейства, как они классифицированы в настоящее время. Лютеовирусы, по-видимому, представляют собой два семейства, а не одно, а apple chlorotic leaf spot virus, по-видимому, является не Closterovirus, а новым родом Potexviridae.
- Эволюция
Эволюция пикорнавирусов, основанная на анализе их РНК-полимераз и геликаз, по-видимому, на сегодняшний день ведет свое происхождение от супергруппы эукариот.[18]. Их предполагаемые предки включают бактериальные ретроэлементы группы II, семейство протеаз HtrA и ДНК-бактериофаги.
Partitiviruses связаны и, возможно, произошли от предка тотивируса.[19]
Гиповирусы и барнавирусы, по-видимому, имеют общую родословную с линиями потивирусов и собемовирусов соответственно.[19]
Двуцепочечные РНК-вирусы
[править | править код]Анализ также предполагает, что дцРНК-вирусы не связаны друг с другом, а вместо этого принадлежат к четырём дополнительным классам — Birnaviridae, Cystoviridae, Partitiviridae и Reoviridae — и одному дополнительному отряду (Totiviridae), одного из классов оц(+)РНК-вирусов и относятся к той же группе, что и РНК-вирусы с положительной цепью.
Одно исследование показало, что существует две большие клады: одна включает семейства Caliciviridae, Flaviviridae и Picornaviridae, а вторая — семейства Alphatetraviridae, Birnaviridae и Cystoviridae, Nodaviridae и Permutotretraviridae.[20]
Одноцепочечные РНК-вирусы с негативной цепью
[править | править код]Эти вирусы имеют сегментированный геном, начиная от одного до восьми сегментов РНК. Несмотря на их разнообразие, они, возможно, возникли у предка членистоногих и дивергировали вместе с ним.[21]
Вирусы-сателлиты
[править | править код]Также известен ряд вирусов-сателлитов — вирусов, для завершения жизненного цикла которых требуется помощь другого вируса. Их таксономия ещё не согласована. Следующие четыре рода были предложены для сателлитных одноцепочечных РНК-вирусов со смысловой цепью, которые заражают растения: Albetovirus, Aumaivirus, Papanivirus и Virtovirus[22]. Семейство Sarthroviridae, включающее род Macronovirus — были предложены для сателлитных одноцепочечных РНК-вирусов со смысловой цепью, которые заражают членистоногих.
Группа III — дцРНК-вирусы
[править | править код]В этой группе насчитывается двенадцать семейств, а также ряд неклассифицированных родов и видов[7].
- Семейство Amalgaviridae
- Семейство Birnaviridae
- Семейство Chrysoviridae
- Семейство Cystoviridae
- Семейство Endornaviridae
- Семейство Hypoviridae
- Семейство Megabirnaviridae
- Семейство Partitiviridae
- Семейство Picobirnaviridae
- Семейство Reoviridae—includes Rotavirus
- Семейство Totiviridae
- Семейство Quadriviridae
- Род Botybirnavirus
- Неклассифицированные роды
Группа IV — оц(+)РНК-вирусы
[править | править код]В этой группе есть три порядка и 34 семейства. Кроме того, существует ряд неклассифицированных и родов.
- Порядок Nidovirales
- Семейство Arteriviridae
- Семейство Coronaviridae — включает Coronavirus (SARS, MERS-CoV, SARS-CoV, SARS-CoV-2)
- Семейство Mesoniviridae
- Семейство Roniviridae
- Порядок Picornavirales
- Семейство Dicistroviridae
- Семейство Iflaviridae
- Семейство Marnaviridae
- Семейство Picornaviridae — включает Poliovirus, Rhinovirus (общая простуда), вирус гепатита A
- Семейство Secoviridae включает подсемейство Comovirinae
- Род Bacillariornavirus
- Род Dicipivirus
- Род Labyrnavirus
- Род Sequiviridae
- Вид Kelp fly virus
- Порядок Tymovirales
- Семейство Alphaflexiviridae
- Семейство Betaflexiviridae
- Семейство Gammaflexiviridae
- Семейство Tymoviridae
- Неклассифицированные
- Семейство Alphatetraviridae
- Семейство Alvernaviridae
- Семейство Astroviridae
- Семейство Barnaviridae
- Семейство Benyviridae
- Семейство Bromoviridae
- Семейство Caliciviridae — включает Norovirus
- Семейство Carmotetraviridae
- Семейство Closteroviridae
- Семейство Flaviviridae — включает вирус жёлтой лихорадки, вирус лихорадки Западного Нила, вирус гепатита C, вирус лихорадки денге, вирус Зика
- Семейство Fusariviridae
- Семейство Hepeviridae
- Семейство Hypoviridae
- Семейство Leviviridae
- Семейство Luteoviridae — включает Barley yellow dwarf virus
- Семейство Polycipiviridae
- Семейство Narnaviridae
- Семейство Nodaviridae
- Семейство Permutotetraviridae
- Семейство Potyviridae
- Семейство Sarthroviridae
- Семейство Statovirus
- Семейство Togaviridae — включает вирус краснухи, Ross River virus, Sindbis virus, Вирус Чикунгунья
- Семейство Tombusviridae
- Семейство Virgaviridae[23]
- Неклассифицированные роды
- Род Blunervirus
- Род Cilevirus
- Род Higrevirus
- Род Idaeovirus
- Род Negevirus
- Род Ourmiavirus
- Род Polemovirus
- Род Sinaivirus
- Род Sobemovirus
- Неклассифицированные виды
- Acyrthosiphon pisum virus
- Bastrovirus
- Blackford virus
- Blueberry necrotic ring blotch virus
- Cadicistrovirus
- Chara australis virus
- Extra small virus
- Goji berry chlorosis virus
- Hepelivirus
- Jingmen tick virus
- Le Blanc virus
- Nedicistrovirus
- Nesidiocoris tenuis virus 1
- Niflavirus
- Nylanderia fulva virus 1
- Orsay virus
- Osedax japonicus RNA virus 1
- Picalivirus
- Plasmopara halstedii virus
- Rosellinia necatrix fusarivirus 1
- Santeuil virus
- Secalivirus
- Solenopsis invicta virus 3
- Wuhan large pig roundworm virus
Вирусы-сателлиты
- Семейство Sarthroviridae
- Род Albetovirus
- Род Aumaivirus
- Род Papanivirus
- Род Virtovirus
- Chronic bee paralysis virus
Также был описан неклассифицированный астровирусный / гепевирусоподобный вирус.[24]
Группа V— оц(-)РНК-вирусы
[править | править код]За исключением гепатита D, эта группа вирусов была помещена в один тип — Negarnaviricota. Этот тип был разделен на два подтипа — Haploviricotina и Polyploviricotina. Внутри подтипа Haploviricotina в настоящее время признаются четыре класса: Chunqiuviricetes, Milneviricetes, Monjiviricetes и Yunchangviricetes. В подтипе Polyploviricotina выделяются два класса: Ellioviricetes и Insthoviricetes.
В настоящее время в этой группе признано 6 классов, 7 порядков и 24 семейства. Чаксть вилов и родов еще не классифицированы.[7]
- Тип Negarnaviricota[25]
- Подтип Haploviricotina
- Класс Chunqiuviricetes
- Порядок Muvirales
- Семейство Qinviridae
- Порядок Muvirales
- Класс Milneviricetes
- Порядок Serpentovirales
- Семейство Aspiviridae
- Порядок Serpentovirales
- Класс Monjiviricetes
- Порядок Jingchuvirales
- Семейство Chuviridae
- Порядок Mononegavirales
- Семейство Bornaviridae — Borna disease virus
- Семейство Filoviridae — включая вирус Эбола, вирус Марбург
- Семейство Mymonaviridae
- Семейство Nyamiviridae[26]
- Семейство Paramyxoviridae — включая вирус кори, Mumps virus, вирус Нипах, Hendra virus, и вирус болезни Ньюкасла
- Семейство Pneumoviridae — включая респираторно-синцитиальный вирус человека и Metapneumovirus
- Семейство Rhabdoviridae — включая вирус бешенства
- Семейство Sunviridae
- Род Anphevirus
- Род Arlivirus
- Род Chengtivirus
- Род Crustavirus
- Род Wastrivirus
- Порядок Jingchuvirales
- Класс Yunchangviricetes
- Порядок Goujianvirales
- Семейство Yueviridae
- Порядок Goujianvirales
- Класс Chunqiuviricetes
- Подтип Polyploviricotina
- Класс Ellioviricetes
- Порядок Bunyavirales
- Семейство Arenaviridae — включая Lassa virus
- Семейство Cruliviridae
- Семейство Feraviridae
- Семейство Fimoviridae
- Семейство Hantaviridae
- Семейство Jonviridae
- Семейство Nairoviridae
- Семейство Peribunyaviridae
- Семейство Phasmaviridae
- Семейство Phenuiviridae
- Семейство Tospoviridae
- Род Tilapineviridae
- Порядок Bunyavirales
- Класс Insthoviricetes
- Порядок Articulavirales
- Семейство Amnoonviridae — включая Taastrup virus
- Семейство Orthomyxoviridae — включая вирусы гриппа
- Порядок Articulavirales
- Класс Ellioviricetes
- Подтип Haploviricotina
- Неклассифицированные роды:
- Род Deltavirus — включая гепатита D
См. также
[править | править код]Примечания
[править | править код]Большинство вирусов, поражающих грибы, представляют собой двухцепочечные РНК-вирусы. Описано небольшое количество одноцепочечных РНК-вирусов с положительной цепью. В одном сообщении высказывается предположение о возможности заражения одноцепочечным вирусом с негативной цепью.[27]
- ↑ MeSH Архивная копия от 16 июня 2010 на Wayback Machine, retrieved on 12 April 2008.
- ↑ Listing in Taxonomic Order—Index to ICTV Species Lists. Дата обращения: 11 апреля 2008. Архивировано из оригинала 6 ноября 2009 года.
- ↑ Drake J. W., Holland J. J. Mutation rates among RNA viruses (англ.) // Proceedings of the National Academy of Sciences of the United States of America : journal. — 1999. — November (vol. 96, no. 24). — P. 13910—13913. — doi:10.1073/pnas.96.24.13910. — . — PMID 10570172. — PMC 24164.
- ↑ Nguyen M., Haenni A. L. Expression strategies of ambisense viruses (неопр.) // Virus Res.. — 2003. — Т. 93, № 2. — С. 141—150. — doi:10.1016/S0168-1702(03)00094-7. — PMID 12782362.
- ↑ Sanjuan, R.; Nebot, M. R.; Chirico, N.; Mansky, L. M.; Belshaw, R. Viral Mutation Rates (англ.) // Journal of Virology[англ.] : journal. — 2010. — Vol. 84, no. 19. — P. 9733—9748. — ISSN 0022-538X. — doi:10.1128/JVI.00694-10. — PMID 20660197. — PMC 2937809.
- ↑ 1 2 3 Klein, Donald W.; Prescott, Lansing M.; Harley, John. Microbiology (неопр.). — Dubuque, Iowa: Wm. C. Brown, 1993. — ISBN 978-0-697-01372-9.
- ↑ Steinhauer D. A., Holland J. J. Rapid evolution of RNA viruses (англ.) // Annu. Rev. Microbiol.. — 1987. — Vol. 41. — P. 409—433. — doi:10.1146/annurev.mi.41.100187.002205. — PMID 3318675.
- ↑ Boutwell C. L., Rolland M. M., Herbeck J. T., Mullins J. I., Allen T. M. Viral Evolution and Escape during Acute HIV-1 Infection (англ.) // J. Infect. Dis. : journal. — 2010. — October (vol. 202, no. Suppl 2). — P. S309—14. — doi:10.1086/655653. — PMID 20846038. — PMC 2945609.
- ↑ Bukh J., Purcell R. H., Miller R. H. Sequence analysis of the core gene of 14 hepatitis C virus genotypes (англ.) // Proceedings of the National Academy of Sciences of the United States of America : journal. — 1994. — August (vol. 91, no. 17). — P. 8239—8243. — doi:10.1073/pnas.91.17.8239. — . — PMID 8058787. — PMC 44581.
- ↑ Tuplin A., Evans D. J., Simmonds P. Detailed mapping of RNA secondary structures in core and NS5B-encoding region sequences of hepatitis C virus by RNase cleavage and novel bioinformatic prediction methods (англ.) // Journal of General Virology[англ.] : journal. — Microbiology Society[англ.], 2004. — October (vol. 85, no. Pt 10). — P. 3037—3047. — doi:10.1099/vir.0.80141-0. — PMID 15448367.
- ↑ Wolf YI, Kazlauskas D, Iranzo J, Lucía-Sanz A, Kuhn JH, Krupovic M, Dolja VV, Koonin EV (2018) Origins and Evolution of the Global RNA Virome. MBio 9(6) pii: e02329-18
- ↑ Francki, R.I.B; Fauquet, C. M.; Knudson, D. L.; Brown, F. Classification and nomenclature of viruses. Fifth report of the International Committee on Taxonomy of Viruses, Archives of Virology (Suppl. 2) (англ.). — 1991. — ISBN 978-3-7091-9163-7.
- ↑ Koonin E. V. The phylogeny of RNA-dependent RNA polymerases of positive-strand RNA viruses (англ.) // Journal of General Virology[англ.] : journal. — Microbiology Society[англ.], 1991. — September (vol. 72, no. Pt 9). — P. 2197—2206. — doi:10.1099/0022-1317-72-9-2197. — PMID 1895057.
- ↑ Rozanov M. N., Koonin E. V., Gorbalenya A. E. Conservation of the putative methyltransferase domain: a hallmark of the 'Sindbis-like' supergroup of positive-strand RNA viruses (англ.) // Journal of General Virology[англ.] : journal. — Microbiology Society[англ.], 1992. — August (vol. 73, no. Pt 8). — P. 2129—2134. — doi:10.1099/0022-1317-73-8-2129. — PMID 1645151.
- ↑ Koonin E. V., Dolja V. V. Evolution and taxonomy of positive-strand RNA viruses: implications of comparative analysis of amino acid sequences (англ.) // Crit. Rev. Biochem. Mol. Biol.[англ.] : journal. — 1993. — Vol. 28, no. 5. — P. 375—430. — doi:10.3109/10409239309078440. — PMID 8269709.
- ↑ Ward C. W. Progress towards a higher taxonomy of viruses (неопр.) // Res Virol. — 1993. — Т. 144, № 6. — С. 419—453. — doi:10.1016/S0923-2516(06)80059-2. — PMID 8140287.
- ↑ Koonin E. V., Wolf Y. I., Nagasaki K., Dolja V. V. The Big Bang of picorna-like virus evolution antedates the radiation of eukaryotic supergroups (англ.) // Nat. Rev. Microbiol. : journal. — 2008. — December (vol. 6, no. 12). — P. 925—939. — doi:10.1038/nrmicro2030. — PMID 18997823.
- ↑ 1 2 Ghabrial SA (1998) Origin, adaptation and evolutionary pathways of fungal viruses. Virus Genes 16(1):119-131
- ↑ Gibrat J. F., Mariadassou M., Boudinot P., Delmas B. Analyses of the radiation of birnaviruses from diverse host phyla and of their evolutionary affinities with other double-stranded RNA and positive strand RNA viruses using robust structure-based multiple sequence alignments and advanced phylogenetic methods (англ.) // BioMed Central[англ.] : journal. — 2013. — Vol. 13. — P. 154. — doi:10.1186/1471-2148-13-154. — PMID 23865988. — PMC 3724706.
- ↑ Li CX, Shi M, Tian JH, Lin XD, Kang YJ, Chen LJ, Qin XC, Xu J, Holmes EC, Zhang YZ (2015) Unprecedented genomic diversity of RNA viruses in arthropods reveals the ancestry of negative-sense RNA viruses. Elife 4. doi: 10.7554/eLife.05378.
- ↑ Krupovic M., Kuhn J. H., Fischer M. G. (2016). A classification system for virophages and satellite viruses. Arch Virol 161(1): 233—247 doi:10.1007/s00705-015-2622-9.
- ↑ Adams M. J., Antoniw J. F., Kreuze J. Virgaviridae: a new family of rod-shaped plant viruses (англ.) // Arch Virol[англ.] : journal. — 2009. — Vol. 154, no. 12. — P. 1967—1972. — doi:10.1007/s00705-009-0506-6. — PMID 19862474.
- ↑ Pankovics P, Boros Á, Kiss T, Engelmann P, Reuter G (2019) Genetically highly divergent RNA virus with astrovirus-like (5'-end) and hepevirus-like (3'-end) genome organization in carnivorous birds, European roller (Coracias garrulus). Infect Genet Evol
- ↑ Virus Taxonomy: 2018 Release. International Committee on Taxonomy of Viruses. Дата обращения: 13 ноября 2018. Архивировано 20 марта 2020 года.
- ↑ Mihindukulasuriya, K. A.; Nguyen, N. L.; Wu, G.; Huang, H. V.; Travassos da Rosa, A. P.; Popov, V. L.; Tesh, R. B.; Wang, D. Nyamanini and Midway viruses define a novel taxon of RNA viruses in the Порядок Mononegavirales (англ.) // J. Virol.[англ.] : journal. — 2009. — Vol. 83, no. 10. — P. 5109—5116. — doi:10.1128/JVI.02667-08. — PMID 19279111. — PMC 2682064.
- ↑ Kondo, H.; Chiba, S.; Toyoda, K.; Suzuki, N. Evidence for negative-strand RNA virus infection in fungi (англ.) // Virology : journal. — 2012. — Vol. 435, no. 2. — P. 201—209. — doi:10.1016/j.virol.2012.10.002. — PMID 23099204.